现在的位置:主页 > 综合新闻 >
利用MONAI加速医学影像学的深度学习研究
【作者】:网站采编【关键词】:【摘要】:利用MONAI加速医学影像学的深度学习研究 / 程序媛的小世界梦寐mayshine Deep Learning Research in Medical Imaging Using MONAI 医学开放式人工智能网络(MONAI)是一个免费提供、社区支持、基于Pytho
利用MONAI加速医学影像学的深度学习研究 / 程序媛的小世界&梦寐mayshine
Deep Learning Research in Medical Imaging Using MONAI
医学开放式人工智能网络(MONAI)是一个免费提供、社区支持、基于Pythorch的医疗影像学深度学习框架。它为开发训练工作流程提供了领域优化的基础功能。
在4月份发布的gtc2020 alpha版本的基础上,MONAI现在发布了0.2版本,为医学成像研究人员提供了新的功能、示例和研究实现,以加快人工智能开发的创新步伐。有关更多信息,请参阅NVIDIA和伦敦国王学院宣布MONAI医疗保健研究开源AI框架。
为什么是MONAI研究?
MONAI research是MONAI代码库中的一个子模块。其目的是展示研究原型的实施和从最新出版的医学影像学与深度学习示范。研究模块由核心开发团队定期审查和维护。根据良好的软件工程实践,从研究子模块中识别出的可重用组件被集成到MONAI核心模块中。
随着MONAI的灵活性和可用性,设想MONAI research是发布研究代码、增加研究影响、促进开放性和可重复性研究的合适场所。像MONAI中的所有其他子模块一样,欢迎以评论、想法和代码的形式发表意见。
在这篇文章中,讨论了目前已经包含在基于MONAI的实现中的研究出版物,这些出版物解决了医学图像分割中的高级研究问题。MONAI不用于临床。
· COPLE-Net: COVID-19 Pneumonia Lesion Segmentation Network:
LAMP: Large Deep Nets with Automated Model Parallelism for Image Segmentation:
src="https://p0.ssl.img.360kuai.com/t01619e26f1d1439241.jpg">图1. COVID19例肺炎病变的CT表现。扫描(a-c)来自三个不同的病人,红色箭头突出显示了一些病变。扫描(d)显示不同观察者给出的(c)注释。
在COVID-19爆发期间,获得大量精确的肺炎病变像素级注释是一项挑战。本研究主要针对分割作业中的噪音标签学习。
本研究的主要创新之一是增强的深卷积神经网络结构。该体系结构具有以下特点:
它使用最大池化和平均池化的组合来减少下采样过程中的信息丢失。
它使用桥接层来减轻编码器和解码器中特征之间的语义鸿沟。
它在瓶颈处采用了ASPP模块,以更好地处理多个尺度的病变。
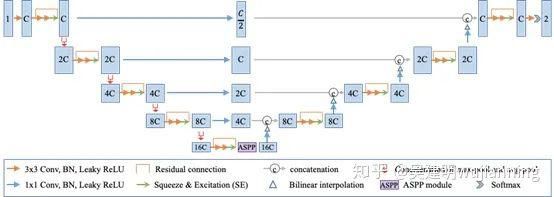 图2. 提出的COPLE网络体系结构。
图2. 提出的COPLE网络体系结构。
这种新颖的建筑是在莫奈提供的。MaxAvgPool和SimpleASPP等关键网络组件可以方便地集成到其他深度学习管道中:
from import MaxAvgPool, SimpleASPP
max_avg_pool = MaxAvgPool(spatial_dims=spatial_dims, kernel_size=2)
aspp = SimpleASPP(spatial_dims, ft_chns[4], int(ft_chns[4] / 4),
kernel_sizes=[1, 3, 3, 3], dilations=[1, 2, 4, 6])
图像预处理管道和预训练模型加载可以通过MONAI的几个Python命令完成:
images = sorted(glob((IMAGE_FOLDER, \"case*.nii.gz\")))
val_files = [{\"img\": img} for img in images]
# define transforms for image and segmentation
infer_transforms = Compose(
[
LoadNiftid(\"img\"),
AddChanneld(\"img\"),
Orientationd(\"img\", \"SPL\"),
ToTensord(\"img\"),
]
test_ds = (data=val_files, transform=infer_transforms)
# sliding window inference need to input one image in every iteration
data_loader = (
test_ds, batch_size=1, num_workers=0, pin_memory=_available()
device = (\"cuda\" if _available() else \"cpu\")
model = CopleNet().to(device)
_state_dict((MODEL_FILE)[\"model_state_dict\"])
Pythorch用户将受益于MONAI医疗图像预处理器和特定领域的网络块。同时,代码显示了MONAI模块和PyTorch本机对象的兼容性,比如,从而简化了MONAI模块在一般PyTorch工作流中的采用。
 图3. 不同损失函数下COPLE网络分割性能的可视化比较。
图3. 不同损失函数下COPLE网络分割性能的可视化比较。
在从噪声标签中学习COVID-19肺炎病灶分割的场景中,COPLE网络的实验结果表明,新的结构比最先进的CNNs具有更高的性能。
LAMP:用于图像分割的自动模型并行的大型深网
深度学习模型变得越来越大,因为模型尺寸的增加可以显著提高精度。通过自动模型并行,可以用大的输入块,甚至是整个图像来训练大型的深3D变换器。
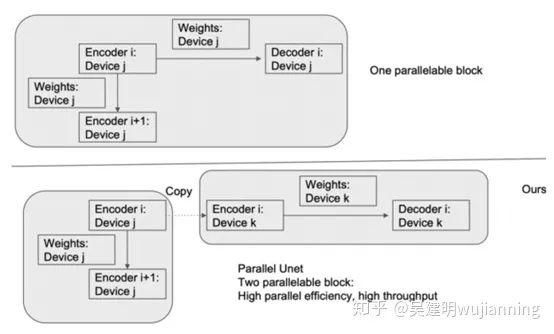 图4. (上)远程跳转连接阻碍了U-Net中的并行性。(底部)显式地构造了U-Net的一个变体,以消除U-Net中的长程依赖。并行U-Net具有较高的并行效率和吞吐量。
图4. (上)远程跳转连接阻碍了U-Net中的并行性。(底部)显式地构造了U-Net的一个变体,以消除U-Net中的长程依赖。并行U-Net具有较高的并行效率和吞吐量。
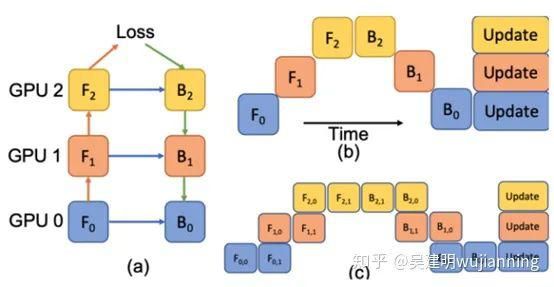 图5. 分区模型。
图5. 分区模型。
在图5中,一个deep模型被划分为三个gpu(a)。Fk是第k细胞的前向功能。Bk是反向传播函数,它依赖于上层的Bk+1和Fk特性。由于模型(b)的依赖性,传统的模型并行性具有较低的设备利用率。流水线并行性将输入的小批量拆分为更小的微批次(c),并允许不同的设备同时运行微批次。同步梯度计算可以最后应用。
文章来源:《医学信息》 网址: http://www.yxxxbjb.cn/zonghexinwen/2020/0803/460.html
上一篇:信息化建设加快 医学影像存档现状如何
下一篇:山西临汾医学院的专业设置

